蛋白质相互作用数据库IID可弥补STRING的不足
蛋白质相互作用数据库IID可弥补STRING的不足
蛋白质作为细胞功能的执行者,主要依赖的是蛋白质之间的相互作用(Protein–protein interactions,PPI),即两种或两种以上的蛋白质结合的过程。研究蛋白相互作用,大家很容易想到的数据库是STRING(https://string-db.org/),STRING虽然知名度高,使用者广泛,但有几点却还是要吐槽一下:
1. 没有区分有实验证据的PPI和基于计算和预测的PPI;
2. 没有细化PPI的属性归类。例如有些PPI是在肌肉组织中检测到的,但数据库统计时则去掉了这一属性,将其推广到在该物种中存在相互作用,而其他组织中可能不存在这个互作关系。
STRING作为一个“粗略、广泛”的搜索工具还是可以的,但是难免混入一些假阳性的结果,这时,IID就是研究路上的另一个重要的科研辅助工具了。
为了解决以上问题,这里介绍一款可用于填坑的研究蛋白互助的数据库IID(http://iid.ophid.utoronto.ca/iid/)

其实,数据库的强大与否,与数据量的关系很大,下图所展示的就是IID所整合的数据。显然,IID数据库涵盖了临床组织标本以及较为全面的动物实验模型的蛋白互助数据,对蛋白互作研究有很大帮助。
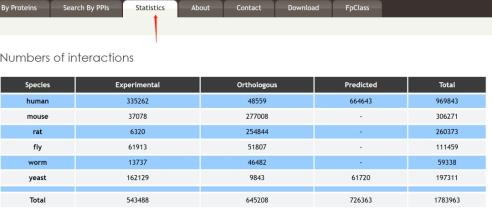
IID主要整合了3种PPI数据:1)通过整合其他9个数据库里有实验证据的PPI包括(BioGRID,IntAct,I2D,MINT,InnateDB,DIP,HPRD,BIND,BCI);2)基于直系同源的PPI;3)从近期的研究里整合具有高可信度的计算预测结果。
IID的功能主要分两部分,一部分是搜索蛋白质的PPI,另一部分是直接检索PPI。下面介绍两部分的使用:
搜索蛋白质的PPI
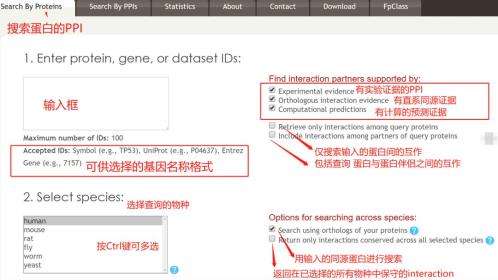
这部分的使用,其实简单粗暴,就是把所有的基因名(注意基因名的格式哦)丢到框框里,然后粗暴的搜索,为了提高搜索结果的准确性,网站作者还提供了一些个性化的设置来帮助大家过滤筛选结果。
笔者再下图中介绍了各个过滤选项的意义,大家可以根据自己的研究目的来选择过滤选项。

直接检索PPI
这部分主要是检索框有差异,其余的部分跟上面的一样。查询差异表达的基因之间是否有相互作用,就可以将基因list丢到这个输入框里,看看这些基因之间是否存在interaction。
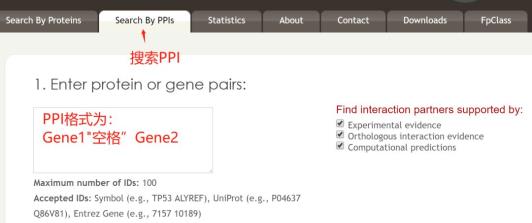
另外,当一个基因的功能明确,要寻找一些调控该基因的其他基因时也可以用这个搜索框帮助大家。然后按照酸菜老师关于基因-基因调控的一元变量研究套路模式代入,就可以找到一些灌水发文的idea啦。
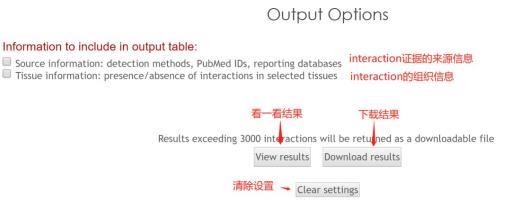
而不论是以第一种搜索方式进行搜索,还是第二种搜索方式,导出的结果格式都是一样的。以TP53为例进行检索,网站会导出结果,格式如下:
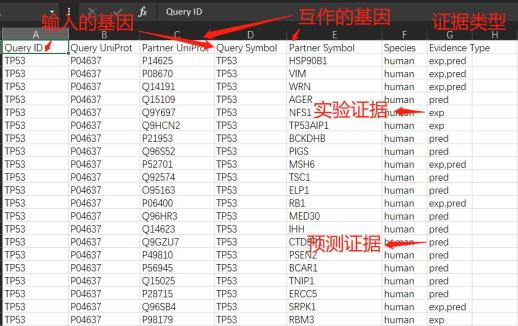
用户可以根据表格的title对结果进行筛选,可以筛选物种、证据类型,证据类型包含了预测的互作证据和实验发现的互作证据。
-
- 2021-03-31
- 2020-07-31
- 2019-09-16
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
-
- 2021-03-31
- 2020-07-31
- 2019-09-16
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
-
- 2021-03-31
- 2020-07-31
- 2019-09-16
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29








