《Nature》端粒“长寿开关”-端粒新结构的发现
在人体的每个细胞中,都有携带决定人体特征的基因的染色体。这些染色体的末端是端粒,可保护染色体免受损伤,它们有点像鞋带末端的塑料头。
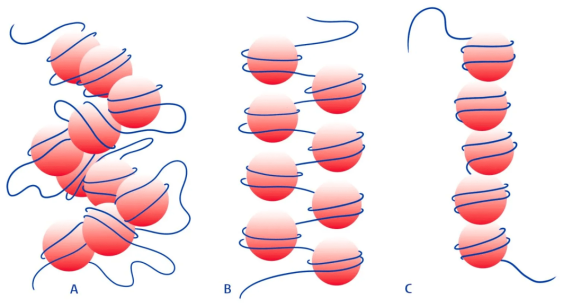
端粒之间的DNA有两米长,因此必须将其折叠以适合细胞。这是通过将DNA包裹在蛋白质上来实现的,DNA和蛋白质一起被称为核小体,它们排列成类似于一串珠子的东西。
在人体的每个细胞中,都有携带决定人体特征的基因的染色体。而端粒就位于这些染色体的末端,用于保护染色体免受损伤。
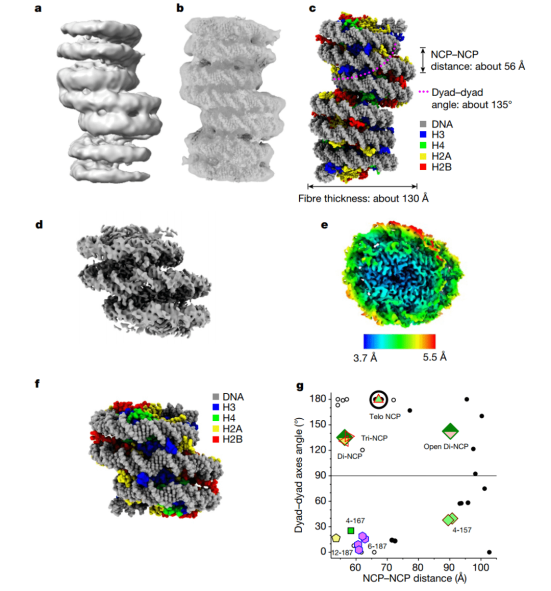
结合电子显微镜和分子力光谱法,荷兰莱顿物理研究所研究人员发现了一种新的端粒结构:核小体之间靠得更近,而它们之间不再有任何游离 DNA。这种结构最终将会产生一个大的 DNA 螺旋。
结合电子显微镜和分子力光谱法,荷兰莱顿物理研究所研究人员发现了另一种端粒结构。在这里,核小体靠得更近,因此珠子之间不再有任何游离DNA。这最终会产生一个大的DNA螺旋。
分子力光谱法将DNA的一端附着在载玻片上,而另一端则粘着一个微小的磁球。然后在这个球上方的一组强磁铁将“珍珠串”拉开。通过测量将珠子一个个拉开所需的力,就可了解更多关于“绳子”如何折叠的信息。
Nature 2022 Sep;609(7929):1048-1055. doi: 10.1038/s41586-022-05236-5.
Columnar structure of human telomeric chromatin
넶浏览量:0
-
- 2021-03-31
- 2020-07-31
- 2019-09-16
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
-
- 2021-03-31
- 2020-07-31
- 2019-09-16
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
-
- 2021-03-31
- 2020-07-31
- 2019-09-16
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29
- 2019-08-29